今天凌晨,微软首席执行官SatyaNadella分享了,微软最新突破性成果蛋白质模拟模型BioEmu。
BioEmu可以将蛋白质动态模拟从数年压缩至几小时,极大加速了医药、生物研究的效率,为个性化医疗带来革命性产品。
目前,微软这项重要研究已经在全球顶刊《自然》上发表。
有网友表示,祝贺你们在AI领域取得了突破性技术成就。希望在不久的将来,在你充满活力的领导下,能看到更多这样的成果。

这是一个重大技术突破。但许多关键的蛋白质功能依赖于非平衡动力学和构象转变。对于人工智能而言,捕捉这些过程仍是一项艰巨的挑战。
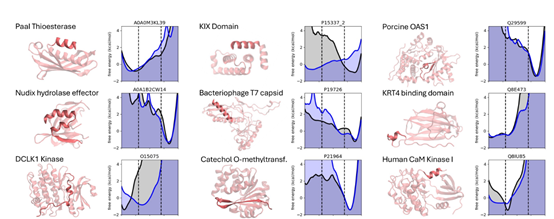
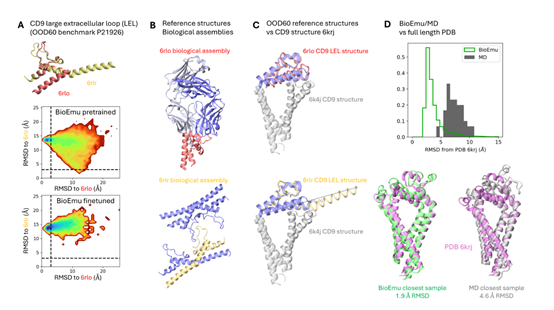
BioEmu能在数小时而非数年内快速追踪蛋白质运动分析,这必将给生物学和药物研发领域带来革命性变革。让我们来看看它对现实世界的应用案例产生了怎样的影响。
BioEmu的发布标志着生物学研究领域的一次变革性飞跃,有望通过以前所未有的速度和精度解析蛋白质动态来重新定义药物研发,这会给医疗健康领域带来革命性变化。

这是一项令人难以置信的突破!将原本需要数年时间的工作缩短至数小时,这确实可能改变药物研发和生物技术领域的格局。这是科学和医疗健康领域的一大步!
BioEmu简单介绍
传统的蛋白质结构解析方法如X射线晶体学和核磁共振技术虽然能够提供高精度的结构信息,但这些方法往往耗时长、成本高,并且对于一些难以结晶或复杂的蛋白质系统效果不佳。
虽然谷歌DeepMind发布的AlphaFold2模型在单个蛋白质结构预测方面取得了巨大成功,但在模拟蛋白质动态平衡构象集合方面仍存在不足。而微软的BioEmu可有效解决这些难题。
BioEmu的核心设计是将蛋白质的序列信息转化为其三维结构的多种可能构象。首先,BioEmu采用了蛋白质序列编码器,这一部分是基于预训练的AlphaFold2模型构建的。AlphaFold2是一个在蛋白质结构预测领域取得重大突破的模型,它通过多序列比对和注意力机制,能够生成高质量的蛋白质序列嵌入。
这些嵌入包含了丰富的进化信息和结构特征,为后续的构象生成提供了坚实的基础。在BioEmu中,序列编码器的作用是将输入的蛋白质序列转化为单个氨基酸和氨基酸对的表示,这些表示将作为后续模块的输入,用于指导蛋白质构象的生成。
接下来是粗粒化蛋白质结构表示模块。为了高效地处理蛋白质的三维结构,BioEmu采用了粗粒化的方法,仅保留蛋白质主链的重原子,即Cα、C、N和O。这种简化不仅减少了计算复杂度,还保留了蛋白质结构的关键信息。
模型通过Gram-Schmidt正交化方法,将每个残基的Cα原子坐标转换为局部坐标系,从而生成蛋白质的主链框架表示。这种表示方式使得模型能够更高效地处理复杂的蛋白质结构,同时保持了对蛋白质动态行为的捕捉能力。
然后是扩散条件生成模型,这是BioEmu的核心部分。扩散模型从一个简单的噪声分布开始,逐步去除噪声,最终生成与目标分布接近的蛋白质构象。这一过程通过分数匹配技术实现,即学习数据的概率分布的梯度,从而指导去噪过程。扩散模型的一个重要特点是它能够生成多样化的构象,这对于捕捉蛋白质的动态行为至关重要。
最后是得分模型,它在BioEmu的架构里扮演着关键角色,主要接收蛋白质序列的单体和对表示、损坏的帧、相对序列位置以及扩散时间步等多方面信息,并据此预测得分。
其结构类似于AlphaFold2和Distributional Graphormer模型的结构模块。值得一提的是,得分模型所生成的平移和旋转得分是在每个残基的局部坐标系中进行定义的,这使得其对整个结构的旋转或平移保持不变。
如此一来,在去噪过程中,骨架原子位置的更新对于整个结构的旋转和平移具备等变性,有力地保障了模型预测的准确性和稳定性。
训练方法
在训练方法上,BioEmu整合了多种异构数据源,其中包括超过200毫秒的分子动力学模拟数据以及实验测量的蛋白质稳定性数据等。在数据处理时,针对不同来源的数据进行了细致的筛选和聚类操作。
以对AFDB数据处理为例,先是使用mmseqs对所有序列在80%序列一致性和70%覆盖率的条件下进行聚类,得到超过9300万个聚类;接着以30%序列一致性对这些聚类的中心点再次聚类,仅保留包含每个30%序列一致性聚类中心点的80%序列一致性聚类;
然后剔除成员少于10个的序列聚类,剩余约140万个序列聚类;之后在每个序列聚类内进行基于结构的聚类,仅保留每个结构聚类的代表成员,同时剔除只有一个结构代表或所有结构代表都是无序结构的序列聚类。
对于分子动力学数据,研究团队广泛收集了公开可用的和内部生成的MD轨迹,模拟时间总计超过200毫秒,涉及24,219条独立链。
此外,BioEmu采用了多阶段训练策略以增强模型的稳定性。在预训练阶段,借助处理后的AFDB数据,通过随机选择序列聚类内的结构,促使模型学习蛋白质构象的多样性;
在微调阶段,基于预训练模型,运用95%的MD模拟数据和蛋白质折叠自由能测量数据,并混合5%的AFDB结构来进行微调。为有效解决MD数据中的采样问题,研究团队运用马尔可夫状态模型、实验数据进行重新加权,以此更好地呈现平衡态分布。
为了高效利用实验测量的蛋白质稳定性数据,BioEmu还开发了一种名为属性预测微调的新颖方法,该方法通过使用差分可训练的目标函数、交叉目标匹配损失项、与常规得分匹配联合训练、梯度累积、使用高阶采样器减少积分时间步、外推以及部分反向传播等一系列技术手段,成功减轻了因过度优化属性预测损失函数而导致的模式崩溃问题,同时大幅降低了计算成本,使得直接反向传播切实可行。
(文:AIGC开放社区)

